ജീനോം (ജീവശാസ്ത്രം)
| Part of a series on |
| Genetics |
|---|
 |
| Key components |
| History and topics |
| Research |
| Personalized medicine |
| Personalized medicine |
| Part of a series on |
| Genetics |
|---|
 |
| Key components |
| History and topics |
| Research |
| Personalized medicine |
| Personalized medicine |

തന്മാത്രാ ജീവശാസ്ത്രത്തിന്റെയും ജനിതകശാസ്ത്രത്തിന്റെയും മേഖലകളിൽ, ഒരു ജീവിയുടെ എല്ലാ ജനിതക വിവരങ്ങളെയും ഒരുമിച്ച് വിളിക്കുന്നത് ആണ് ജീനോം എന്നത്.[1] ഡിഎൻഎയുടെ ന്യൂക്ലിയോടൈഡ് സീക്വൻസുകൾ ഇതിൽ അടങ്ങിയിരിക്കുന്നു (അല്ലെങ്കിൽ ആർഎൻഎ വൈറസുകളിലെ ആർഎൻഎ). ന്യൂക്ലിയർ ജീനോമിൽ പ്രോട്ടീൻ-കോഡിംഗ് ജീനുകൾ, നോൺ-കോഡിംഗ് ജീനുകൾ, ജീനോമിന്റെ മറ്റ് പ്രവർത്തന മേഖലകളായ റെഗുലേറ്ററി സീക്വൻസുകൾ (നോൺ-കോഡിംഗ് ഡിഎൻഎ കാണുക), പലപ്പോഴും വ്യക്തമായ പ്രവർത്തനങ്ങളില്ലാത്ത ജങ്ക് ഡിഎൻഎകളുടെ ഭാഗങ്ങൾ എന്നിവ അടങ്ങിയിരിക്കുന്നു.[2][3] മിക്കവാറും എല്ലാ യൂക്കാരിയോട്ടുകൾക്കും മൈറ്റോകോൺഡ്രിയയും ഒരു ചെറിയ മൈറ്റോകോൺഡ്രിയൽ ജീനോമും ഉണ്ട്.[2] ആൽഗകളിലും സസ്യങ്ങളിലും ക്ലോറോപ്ലാസ്റ്റ് ജീനോം ഉള്ള ക്ലോറോപ്ലാസ്റ്റുകളും അടങ്ങിയിട്ടുണ്ട്.
ജീനോമിനെക്കുറിച്ചുള്ള പഠനത്തെ ജീനോമിക്സ് എന്ന് വിളിക്കുന്നു. പല ജീവികളുടെയും ജീനോമുകൾ സീക്വൻസ് നടത്തി അതിലെ വിവിധ പ്രദേശങ്ങൾ വ്യാഖ്യാനിക്കപ്പെട്ടിട്ടുണ്ട്. 1990 ഒക്ടോബറിൽ ഹ്യൂമൻ ജീനോം പ്രോജക്റ്റ് ആരംഭിച്ചു, തുടർന്ന് 2003 ഏപ്രിലിൽ ഹ്യൂമൻ ജീനോമിന്റെ സീക്വൻസ് റിപ്പോർട്ട് ചെയ്തു,[4] പ്രാരംഭ ഘട്ടത്തിലെ "പൂർത്തിയായ" ശ്രേണിയിൽ ഭൂരിഭാഗവും ആവർത്തന ശ്രേണികൾ അടങ്ങിയ ജീനോമിന്റെ 8% കാണുന്നില്ല.[5]
നിരവധി ആവർത്തന ശ്രേണികളുടെ ക്രമം കൈകാര്യം ചെയ്യാൻ കഴിയുന്ന സാങ്കേതികവിദ്യയിലെ പുരോഗതിയോടെ, ശാസ്ത്രജ്ഞർ, ആദ്യത്തെ യഥാർത്ഥ ഹ്യൂമൻ ജീനോം പ്രോജക്ട് പഠനത്തിലൂടെ പൂർണ്ണമായി കണ്ടെത്താത്ത ആവർത്തന സീക്വൻസുകൾ ഉൾപ്പടെയുള്ള ആദ്യത്തെ മനുഷ്യ ജീനോം സീക്വൻസ് റിപ്പോർട്ട് 2022 മാർച്ചിൽ പുറത്തിറക്കി.[6]
പദോൽപ്പത്തി
[തിരുത്തുക]ജർമ്മനിയിലെ ഹാംബർഗ് സർവ്വകലാശാലയിലെ സസ്യശാസ്ത്ര പ്രൊഫസറായ ഹാൻസ് വിങ്ക്ലർ ആണ് 1920-ൽ ജീനോം എന്ന പദം സൃഷ്ടിച്ചത്.[7] ഓക്സ്ഫോർഡ് നിഘണ്ടു എന്ന വെബ്സൈറ്റും ഓൺലൈൻ എറ്റിമോളജി നിഘണ്ടുവും സൂചിപ്പിക്കുന്നത് ജീൻ, ക്രോമസോം എന്നീ പദങ്ങളുടെ മിശ്രിതമാണ് ഈ പേര് എന്നാണ്.[8][9] എന്നിരുന്നാലും, കൂടുതൽ സമഗ്രമായ ചർച്ചയ്ക്ക് ഒമിക്സ് കാണുക. ബയോം, റൈസോം എന്നിവ പോലുള്ള ചില അനുബന്ധ -ഓം പദങ്ങൾ അതിനകം നിലവിലുണ്ടായിരുന്നു.[10]
നിർവ്വചനം
[തിരുത്തുക]"ജീനോം" എന്നതിന്റെ കൃത്യമായ നിർവചനം പറയുന്നത് വളരെ ബുദ്ധിമുട്ടാണ്. ഇത് സാധാരണയായി ഒരു ജീവിയിലെ ജനിതക വിവരങ്ങൾ വഹിക്കുന്ന ഡിഎൻഎ (അല്ലെങ്കിൽ ചിലപ്പോൾ ആർഎൻഎ) തന്മാത്രകളെയാണ് സൂചിപ്പിക്കുന്നത്, എന്നാൽ നിർവചനത്തിൽ ഏതൊക്കെ തന്മാത്രകൾ ഉൾപ്പെടുത്തണമെന്ന് തീരുമാനിക്കുന്നത് ചിലപ്പോൾ ബുദ്ധിമുട്ടാണ്; ഉദാഹരണത്തിന്, ബാക്ടീരിയകൾക്ക് സാധാരണയായി ഒന്നോ രണ്ടോ വലിയ ഡിഎൻഎ തന്മാത്രകൾ (ക്രോമസോമുകൾ) ഉണ്ടായിരിക്കും, അവയിൽ എല്ലാ അവശ്യ ജനിതക വസ്തുക്കളും അടങ്ങിയിരിക്കുന്നു, എന്നാൽ അവയിൽ പ്രധാനപ്പെട്ട ജനിതക വിവരങ്ങൾ വഹിക്കുന്ന ചെറിയ എക്സ്ട്രാക്രോമസോമൽ പ്ലാസ്മിഡ് തന്മാത്രകളും അടങ്ങിയിരിക്കുന്നു. ശാസ്ത്രസാഹിത്യത്തിൽ സാധാരണയായി ഉപയോഗിക്കുന്ന 'ജീനോം' എന്നതിന്റെ നിർവചനം സാധാരണയായി ബാക്ടീരിയയിലെ വലിയ ക്രോമസോം ഡിഎൻഎ തന്മാത്രകളിലേക്ക് പരിമിതപ്പെടുത്തിയിരിക്കുന്നു. [11]
യൂക്കാരിയോട്ടിക് ജീനോമുകൾ നിർവചിക്കാൻ കൂടുതൽ ബുദ്ധിമുട്ടാണ്, കാരണം മിക്കവാറും എല്ലാ യൂക്കാരിയോട്ടിക് സ്പീഷീസുകളിലും ന്യൂക്ലിയർ ക്രോമസോമുകളും മൈറ്റോകോണ്ട്രിയയിലെ അധിക ഡിഎൻഎ തന്മാത്രകളും അടങ്ങിയിരിക്കുന്നു. കൂടാതെ, ആൽഗകൾക്കും സസ്യങ്ങൾക്കും ക്ലോറോപ്ലാസ്റ്റ് ഡിഎൻഎ ഉണ്ട്. മിക്ക പാഠപുസ്തകങ്ങളും ന്യൂക്ലിയർ ജീനോമും ഓർഗനെല്ല (മൈറ്റോകോൺഡ്രിയയും ക്ലോറോപ്ലാസ്റ്റും) ജീനോമും തമ്മിൽ വ്യത്യാസം കാണിക്കുന്നു, അതിനാൽ മനുഷ്യ ജീനോമിനെക്കുറിച്ച് പറയുമ്പോൾ, അവ ന്യൂക്ലിയസിലെ ജനിതക വസ്തുക്കളെ മാത്രമേ പരാമർശിക്കുന്നുള്ളൂ. [2] [12] ശാസ്ത്രസാഹിത്യത്തിൽ 'ജീനോമിന്റെ' ഏറ്റവും സാധാരണമായ ഉപയോഗമാണിത്.
മിക്ക യൂക്കാരിയോട്ടുകളും ഡിപ്ലോയിഡ് ആണ്, അതായത് ന്യൂക്ലിയസിൽ ഓരോ ക്രോമസോമിന്റെയും രണ്ട് പകർപ്പുകൾ ഉണ്ട്, എന്നാൽ 'ജീനോം' ഓരോ ക്രോമസോമിന്റെയും ഒരു പകർപ്പിനെ മാത്രമേ സൂചിപ്പിക്കുന്നുള്ളൂ. ചില യൂക്കാരിയോട്ടുകൾക്ക് സസ്തനികളുടെ X, Y ക്രോമസോമുകൾ പോലെയുള്ള വ്യതിരിക്തമായ സെക്സ് ക്രോമസോമുകൾ ഉണ്ട്, അതിനാൽ ജീനോമിന്റെ സാങ്കേതിക നിർവചനത്തിൽ ലൈംഗിക ക്രോമസോമുകളുടെ രണ്ട് പകർപ്പുകളും ഉൾപ്പെടുത്തണം. ഉദാഹരണത്തിന്, മനുഷ്യരുടെ സ്റ്റാൻഡേർഡ് റഫറൻസ് ജീനോമിനെ പരാമർശിക്കുമ്പോൾ, അതിൽ 22 ഓട്ടോസോമുകളുടെ ഓരോ പകർപ്പും കൂടാതെ ഒരു X ക്രോമസോമും ഒരു Y ക്രോമസോമും അടങ്ങിയിരിക്കുന്നു. [13]
സീക്വൻസിംഗും മാപ്പിംഗും
[തിരുത്തുക]
ഒരു വ്യക്തിയുടെയോ ഒരു ജീവിവർഗത്തിന്റെയോ എല്ലാ ക്രോമസോമുകളും നിർമ്മിക്കുന്ന ന്യൂക്ലിയോടൈഡുകളുടെ (ഡിഎൻഎ ജീനോമുകൾക്കുള്ള എ, സി, ജി, ടി) പൂർണ്ണമായ പട്ടികയാണ് ജീനോം സീക്വൻസ്. ഒരു സ്പീഷിസിനുള്ളിൽ, ബഹുഭൂരിപക്ഷം ന്യൂക്ലിയോടൈഡുകളും വ്യക്തികൾക്കിടയിൽ സമാനമാണ് എന്നതിനാൽ ജനിതക വൈവിധ്യം മനസ്സിലാക്കാൻ ഒന്നിലധികം വ്യക്തികളെ ക്രമപ്പെടുത്തേണ്ടത് ആവശ്യമാണ്.
1976-ൽ, ഗെൻറ് സർവകലാശാലയിലെ (ബെൽജിയം) വാൾട്ടർ ഫിയേഴ്സ് ആണ് ഒരു വൈറൽ ആർഎൻഎ-ജീനോമിന്റെ (ബാക്ടീരിയോഫേജ് എംഎസ്2) പൂർണ്ണമായ ന്യൂക്ലിയോടൈഡ് സീക്വൻസ് ആദ്യമായി രേഖപ്പെടുത്തുന്നത്. അടുത്ത വർഷം, ഫ്രെഡ് സാംഗർ ആദ്യത്തെ ഡിഎൻഎ-ജീനോം സീക്വൻസ് പൂർത്തിയാക്കി: Phage Φ-X174, 5386 അടിസ്ഥാന ജോഡികൾ.[14] 1995-ൽ ഇൻസ്റ്റിറ്റ്യൂട്ട് ഫോർ ജീനോമിക് റിസർച്ചിലെ ഒരു സംഘം പൂർത്തിയാക്കിയ ഹീമോഫിലസ് ഇൻഫ്ലുവൻസയുടേതാണ് ആദ്യത്തെ സീക്വൻസ് ചെയ്ത ബാക്ടീരിയൽ ജീനോം. ഏതാനും മാസങ്ങൾക്കുശേഷം, 1980-കളുടെ മധ്യത്തിൽ ആരംഭിച്ച യൂറോപ്യൻ നേതൃത്വത്തിലുള്ള ശ്രമത്തിന്റെ ഫലമായി, വളർന്നുവരുന്ന യീസ്റ്റ് സാക്കറോമൈസസ് സെറിവിസിയയുടെ 16 ക്രോമസോമുകളുടെ ക്രമങ്ങൾ പ്രസിദ്ധീകരിച്ച് ആദ്യത്തെ യൂക്കറിയോട്ടിക് ജീനോം പൂർത്തിയായി. മെത്തനോകോക്കസ് ജന്നാസ്ചി എന്ന പുരാവസ്തുവിന്റെ ആദ്യ ജീനോം സീക്വൻസ് 1996-ൽ വീണ്ടും ദ ഇൻസ്റ്റിറ്റ്യൂട്ട് ഫോർ ജീനോമിക് റിസർച്ച് പൂർത്തിയാക്കി.
പുതിയ സാങ്കേതികവിദ്യകളുടെ വികസനം ജീനോം സീക്വൻസിംഗിനെ ചിലവ് കുറഞ്ഞതും എളുപ്പവുമാക്കിത്തീർക്കുന്നു, അതിലൂടെ സമ്പൂർണ്ണ ജീനോം സീക്വൻസുകളുടെ എണ്ണം അതിവേഗം വളരുകയാണ്. യുഎസ് നാഷണൽ ഇൻസ്റ്റിറ്റ്യൂട്ട് ഓഫ് ഹെൽത്ത് ജനിതക വിവരങ്ങളുടെ നിരവധി സമഗ്രമായ ഡാറ്റാബേസുകളിലൊന്ന് പരിപാലിക്കുന്നു. [15] പൂർത്തിയായ ആയിരക്കണക്കിന് ജീനോം സീക്വൻസിംഗ് പ്രോജക്റ്റുകളിൽ അരി, ഒരു എലി, അറബിഡോപ്സിസ് താലിയാന എന്ന സസ്യം, പഫർ ഫിഷ്, ബാക്ടീരിയ ഇ.കോളി എന്നിവ ഉൾപ്പെടുന്നു. 2013 ഡിസംബറിൽ, വംശനാശം സംഭവിച്ച മനുഷ്യവർഗമായ നിയാണ്ടർത്താലിന്റെ മുഴുവൻ ജീനോമും ശാസ്ത്രജ്ഞർ ആദ്യമായി ക്രമീകരിച്ചു. സൈബീരിയൻ ഗുഹയിൽ നിന്ന് കണ്ടെത്തിയ 1,30,000 വർഷം പഴക്കമുള്ള നിയാണ്ടർത്താലിന്റെ കാൽവിരൽ അസ്ഥിയിൽ നിന്നാണ് ജനിതകഘടന വേർതിരിച്ചെടുത്തത്.[16][17]
മാന്തിയ പ്രെഡിക്റ്റീവ് മെഡിസിൻ മുൻകൈയെടുത്തത് പോലെ, മാസിവ് പാരലൽ സീക്വൻസിംഗ് പോലുള്ള പുതിയ സീക്വൻസിംഗ് സാങ്കേതികവിദ്യകൾ ഒരു ഡയഗ്നോസ്റ്റിക് ഉപകരണം എന്ന നിലയിൽ വ്യക്തിഗത ജീനോം സീക്വൻസിംഗിന്റെ സാധ്യതയും തുറന്നു. ആ ലക്ഷ്യത്തിലേക്കുള്ള ഒരു പ്രധാന ചുവടുവയ്പ്പാണ് 2007-ൽ ഡിഎൻഎയുടെ ഘടനയുടെ സഹ-കണ്ടെത്തിയവരിൽ ഒരാളായ ജെയിംസ് ഡി വാട്സന്റെ പൂർണ്ണ ജീനോം പൂർത്തിയാക്കിയത്.[18]
ഒരു ജീനോം സീക്വൻസ് ഒരു ജീനോമിലെ എല്ലാ ഡിഎൻഎ ബേസിന്റെയും ക്രമം പട്ടികപ്പെടുത്തുമ്പോൾ, ഒരു ജീനോം മാപ്പ് ലാൻഡ്മാർക്കുകൾ മാത്രം തിരിച്ചറിയുന്നു. ഒരു ജീനോം മാപ്പിൽ ഒരു ജീനോം സീക്വൻസിനേക്കാൾ വിവരങ്ങൾ കുറവാണ്. മനുഷ്യ ജീനോം മാപ്പ് ചെയ്യുന്നതിനും ക്രമപ്പെടുത്തുന്നതിനുമായി ഹ്യൂമൺ ജീനോം പ്രോജക്റ്റ് ആരംഭിച്ചു. പാരീസിലെ ജെനോസ്കോപ്പിൽ വെച്ച് ജീൻ വെയ്സെൻബാക്കും സംഘവും നടത്തിയ വിശദമായ ജീനോമിക് മാപ്പ് പുറത്തിറക്കിയതാണ് പദ്ധതിയുടെ അടിസ്ഥാന ഘട്ടം.[19][20]
വൈറൽ ജീനോമുകൾ
[തിരുത്തുക]വൈറൽ ജീനോമുകൾ ആർഎൻഎ അല്ലെങ്കിൽ ഡിഎൻഎ അടങ്ങിയതായിരിക്കാം. ആർഎൻഎ വൈറസുകളുടെ ജീനോമുകൾ സിംഗിൾ സ്ട്രാൻഡഡ് ആർഎൻഎ അല്ലെങ്കിൽ ഡബിൾ സ്ട്രാൻഡഡ് ആർഎൻഎ ആകാം, കൂടാതെ ഒന്നോ അതിലധികമോ പ്രത്യേക ആർഎൻഎ തന്മാത്രകൾ അതിൽ അടങ്ങിയിരിക്കാം (സെഗ്മെന്റുകൾ: മോണോപാർട്ടിറ്റ് അല്ലെങ്കിൽ മൾട്ടിപാർട്ടിറ്റ് ജീനോം). ഡിഎൻഎ വൈറസുകൾക്ക് സിംഗിൾ സ്ട്രാൻഡഡ് അല്ലെങ്കിൽ ഡബിൾ സ്ട്രാൻഡഡ് ജീനോമുകൾ ഉണ്ടാകാം. മിക്ക ഡിഎൻഎ വൈറസ് ജീനോമുകളും ഡിഎൻഎയുടെ ഒറ്റ, ലീനിയർ തന്മാത്ര കൊണ്ടാണ് നിർമ്മിച്ചിരിക്കുന്നത്, എന്നാൽ ചിലത് സർക്കുലർ ഡിഎൻഎ തന്മാത്ര കൊണ്ടാണ് നിർമ്മിച്ചിരിക്കുന്നത്.[21]
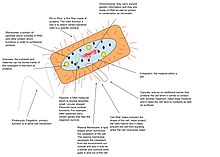
പ്രോകാരിയോട്ടിക് ജീനോമുകൾ
[തിരുത്തുക]പ്രോകാരിയോട്ടുകൾക്കും യൂക്കാരിറിയോട്ടുകൾക്കും ഡിഎൻഎ ജീനോമുകൾ ഉണ്ട്. ആർക്കിയയ്ക്കും മിക്ക ബാക്ടീരിയകൾക്കും ഒരൊറ്റ സർക്കുലർ (വൃത്താകൃതിയിലുള്ള) ക്രോമസോം ആണ് ഉള്ളത്,[22] എന്നാൽ, ചില ബാക്ടീരിയൽ സ്പീഷീസുകൾക്ക് ലീനിയർ ക്രോമോസോം അല്ലെങ്കിൽ ഒന്നിലധികം ക്രോമസോമുകൾ ഉണ്ട്.[23][24] ബാക്ടീരിയൽ കോശങ്ങൾ വിഭജിക്കുന്നതിനേക്കാൾ വേഗത്തിൽ ഡിഎൻഎ പകർത്തിയാൽ, ക്രോമസോമിന്റെ ഒന്നിലധികം പകർപ്പുകൾ ഒരു കോശത്തിൽ ഉണ്ടാകും, ഡിഎൻഎയെക്കാൾ വേഗത്തിൽ കോശങ്ങൾ വിഭജിക്കുകയാണെങ്കിൽ, വിഭജനം സംഭവിക്കുന്നതിന് മുമ്പ് ക്രോമസോമിന്റെ ഒന്നിലധികം പകർപ്പുകൾ ആരംഭിക്കും. മിക്ക പ്രോകാരിയോട്ടുകളുടെയും ജീനോമുകളിൽ ആവർത്തിച്ചുള്ള ഡിഎൻഎ വളരെ കുറവാണ്.[25] എന്നിരുന്നാലും, ചില സിംബയോട്ടിക് ബാക്ടീരിയകൾക്ക് (ഉദാ: സെറാറ്റിയ സിംബയോട്ടിക്ക) കുറഞ്ഞ ജീനോമുകളും സ്യൂഡോജെനുകളുടെ ഉയർന്ന അംശവും കാണാം: അവയുടെ ഡിഎൻഎയുടെ ~40% പ്രോട്ടീനുകളെ എൻകോഡ് ചെയ്യുന്നു.[26][27]
ചില ബാക്ടീരിയകൾക്ക് ഓക്സിലറി ജനിറ്റിക് മെറ്റീരിയൽ ഉണ്ട്, അവയുടെ ജനിതകഘടനയുടെ ഭാഗവും പ്ലാസ്മിഡുകളിൽ വഹിക്കുന്നു. ഇവിടെ, ക്രോമസോമിന്റെ പര്യായമായി ജീനോം എന്ന വാക്ക് ഉപയോഗിക്കരുത്.
യൂക്കാരിയോട്ടിക് ജീനോമുകൾ
[തിരുത്തുക]ഒന്നോ അതിലധികമോ ലീനിയർ ഡിഎൻഎ ക്രോമസോമുകൾ ചേർന്നതാണ് യൂക്കാരിയോട്ടിക് ജീനോമുകൾ. ക്രോമസോമുകളുടെ എണ്ണം, ഒരു ജോഡി മാത്രമുള്ള ജാക്ക് ജമ്പർ ഉറുമ്പുകൾ,[28] മുതൽ 720 ജോഡികളുള്ള ഫേൺ സ്പീഷീസ് വരെ വ്യത്യാസപ്പെടാം.[29] മറ്റ് ജീനോമുകളെ അപേക്ഷിച്ച് യൂക്കാരിയോട്ടിക് ജീനോമുകളിൽ അടങ്ങിയിരിക്കുന്ന ഡിഎൻഎയുടെ അളവ് അതിശയിപ്പിക്കുന്നതാണ്.[30]
ഒരു സാധാരണ മനുഷ്യ കോശത്തിൽ മാതാപിതാക്കളിൽ നിന്നും പാരമ്പര്യമായി ലഭിക്കുന്ന 22 ഓട്ടോസോമുകളുടെ രണ്ട് പകർപ്പുകൾ ഉണ്ട്, കൂടാതെ രണ്ട് ലൈംഗിക ക്രോമസോമുകൾ കൂടി ചേർന്ന് അത് ഡിപ്ലോയിഡ് ആകുന്നു. അണ്ഡം, ബീജം, ബീജങ്ങൾ, കൂമ്പോള എന്നിവ പോലുള്ള ഗാമീറ്റുകൾ ഹാപ്ലോയിഡ് ആണ്, അതായത് ഓരോ ക്രോമസോമിന്റെയും ഒരു പകർപ്പ് മാത്രമേ അവ വഹിക്കുന്നുള്ളൂ. ന്യൂക്ലിയസിലെ ക്രോമസോമുകൾക്ക് പുറമേ, ക്ലോറോപ്ലാസ്റ്റുകൾ, മൈറ്റോകോണ്ട്രിയ തുടങ്ങിയവക്കും അവരുടേതായ ഡിഎൻഎ ഉണ്ട്. മൈറ്റോകോൺഡ്രിയയ്ക്ക് സ്വന്തം ജീനോം ഉണ്ടെന്ന് ചിലപ്പോൾ പറയാറുണ്ട്, ഇതിനെ പലപ്പോഴും "മൈറ്റോകോൺഡ്രിയൽ ജീനോം" എന്ന് വിളിക്കാറുണ്ട്. ക്ലോറോപ്ലാസ്റ്റിനുള്ളിൽ കാണപ്പെടുന്ന ഡിഎൻഎയെ "പ്ലാസ്റ്റോം" എന്ന് വിളിക്കാറുണ്ട്. അവ ഉത്ഭവിച്ച ബാക്ടീരിയകളെപ്പോലെ, മൈറ്റോകോൺഡ്രിയയ്ക്കും ക്ലോറോപ്ലാസ്റ്റുകൾക്കും വൃത്താകൃതിയിലുള്ള സർക്കുലർ ക്രോമസോം ഉണ്ട്.
ഡിഎൻഎ സീക്വൻസിങ്
[തിരുത്തുക]ഒരു ഡിഎൻഎ തന്മാത്രയിലെ ന്യൂക്ലിയോടൈഡുകളുടെ അല്ലെങ്കിൽ ബേസുകളുടെ കൃത്യമായ ക്രമം നിർണ്ണയിക്കുന്നതിനുള്ള പൊതു ലബോറട്ടറി സാങ്കേതികതയാണ് ഡിഎൻഎ സീക്വൻസിങ്. ബേസുകളുടെ ക്രമം (പലപ്പോഴും അവയുടെ രാസനാമങ്ങളുടെ ആദ്യ അക്ഷരങ്ങളാൽ പരാമർശിക്കപ്പെടുന്നു: എ, ടി, സി, ജി) കോശങ്ങൾ വികസിപ്പിക്കുന്നതിനും പ്രവർത്തിക്കുന്നതിനും ഉപയോഗിക്കുന്ന ജൈവ വിവരങ്ങൾ എൻകോഡ് ചെയ്യുന്നു. അനുക്രമമായ ഐസൊലേറ്റുകളെ ഒരു റഫറൻസുമായി താരതമ്യം ചെയ്താണ് സീക്വൻസ് പോളിമോർഫിസങ്ങൾ സാധാരണയായി കണ്ടെത്തുന്നത്, അതേസമയം കവറേജ് ഡെപ്ത്, മാപ്പിംഗ് ടോപ്പോളജി എന്നിവയുടെ വിശകലനങ്ങൾക്ക് ക്രോമസോം ട്രാൻസ്ലോക്കേഷനുകളും സെഗ്മെന്റൽ ഡ്യൂപ്ലിക്കേഷനുകളും പോലുള്ള ഘടനാപരമായ വ്യതിയാനങ്ങളെക്കുറിച്ചുള്ള വിശദാംശങ്ങൾ നൽകാൻ കഴിയും.
കോഡിംഗ് സീക്വൻസുകൾ
[തിരുത്തുക]പ്രോട്ടീനുകൾ നിർമ്മിക്കുന്നതിനുള്ള നിർദ്ദേശങ്ങൾ ഉൾക്കൊള്ളുന്ന ഡിഎൻഎ സീക്വൻസുകളെ കോഡിംഗ് സീക്വൻസുകൾ എന്ന് വിളിക്കുന്നു. കോഡിംഗ് സീക്വൻസുകൾ ഉൾക്കൊള്ളുന്ന ജീനോമിന്റെ അനുപാതം വ്യാപകമായി വ്യത്യാസപ്പെടുന്നു. ഒരു വലിയ ജീനോമിൽ കൂടുതൽ ജീനുകൾ ഉണ്ടായിരിക്കണമെന്നില്ല, സങ്കീർണ്ണമായ യൂക്കാരിയോട്ടുകളിൽ ജീനോം വലുപ്പം വർദ്ധിക്കുന്നതിനൊപ്പം ആവർത്തിക്കാത്ത ഡിഎൻഎയുടെ അനുപാതം കുറയുന്നു.[31]

നോൺകോഡിംഗ് സീക്വൻസുകൾ
[തിരുത്തുക]നോൺകോഡിംഗ് സീക്വൻസുകളിൽ ഇൻട്രോണുകൾ, നോൺ-കോഡിംഗ് ആർഎൻഎകൾക്കുള്ള സീക്വൻസുകൾ, റെഗുലേറ്ററി റീജിയണുകൾ, റെപ്പറ്റീറ്റീവ് ഡിഎൻഎ എന്നിവ ഉൾപ്പെടുന്നു. മനുഷ്യ ജീനോമിന്റെ 98 ശതമാനവും നോൺകോഡിംഗ് സീക്വൻസുകളാണ്. ജീനോമിൽ റെപ്പറ്റീറ്റീവ് ഡിഎൻഎയുടെ രണ്ട് വിഭാഗങ്ങളുണ്ട്: ടാൻഡം റിപ്പീറ്റുകളും ഇന്റർസ്പെഴ്സ്ഡ് റിപ്പീറ്റുകളും.[32]
ജീനോം വലിപ്പം
[തിരുത്തുക]ഒരു ഹാപ്ലോയിഡ് ജീനോമിന്റെ ഒരു പകർപ്പിലെ ഡിഎൻഎ അടിസ്ഥാന ജോഡികളുടെ ആകെ എണ്ണമാണ് ജീനോം വലുപ്പം എന്ന് അറിയപ്പെടുന്നത്. ജീവിവർഗങ്ങളിലുടനീളം ജീനോം വലുപ്പം വ്യാപകമായി വ്യത്യാസപ്പെടുന്നു. അകശേരുക്കൾക്ക് ചെറിയ ജീനോമുകൾ ഉണ്ട്, ഇത് ട്രാൻസ്പോസിബിൾ മൂലകങ്ങളുടെ ഒരു ചെറിയ സംഖ്യയുമായി ബന്ധപ്പെട്ടിരിക്കുന്നു. മത്സ്യങ്ങൾക്കും ഉഭയജീവികൾക്കും ഇടത്തരം വലിപ്പമുള്ള ജീനോമുകൾ ഉണ്ട്, പക്ഷികൾക്ക് താരതമ്യേന ചെറിയ ജീനോമുകൾ ഉണ്ട്, എന്നാൽ പറക്കലിലേക്കുള്ള പരിവർത്തന ഘട്ടത്തിൽ പക്ഷികൾക്ക് അവയുടെ ജീനോമുകളുടെ ഗണ്യമായ ഭാഗം നഷ്ടപ്പെട്ടതായി അഭിപ്രായമുണ്ട്. ഈ നഷ്ടത്തിന് മുമ്പ്, ഡിഎൻഎ മെഥൈലേഷൻ ജീനോമിന്റെ മതിയായ വികാസം അനുവദിക്കുന്നു.[30]
മനുഷ്യരിലെ ന്യൂക്ലിയർ ജീനോമിൽ ഡിഎൻഎയുടെ ഏകദേശം 3.1 ബില്യൺ ന്യൂക്ലിയോടൈഡുകൾ ഉൾക്കൊള്ളുന്നു, അവ 24 ലീനിയർ തന്മാത്രകളായി തിരിച്ചിരിക്കുന്നു, ഇതിൽ നീളം കുറഞ്ഞ 45,000,000 ന്യൂക്ലിയോടൈഡുകൾ, ദൈർഘ്യമേറിയ 248,000,000 ന്യൂക്ലിയോടൈഡുകൾ എന്നിവയുണ്ട്.[33] പ്രോകാരിയോട്ടുകളിലോ ലോവർ യൂക്കാരിയോട്ടുകളിലോ രൂപാന്തര സങ്കീർണ്ണതയും ജീനോം വലുപ്പവും തമ്മിൽ വ്യക്തവും സ്ഥിരവുമായ ബന്ധമില്ല.[31][34] ആവർത്തിച്ചുള്ള ഡിഎൻഎ മൂലകങ്ങളുടെ വികാസത്തിന്റെയും സങ്കോചത്തിന്റെയും പ്രവർത്തനമാണ് ജീനോം വലുപ്പം.
ജീനോമിക് മാറ്റങ്ങൾ
[തിരുത്തുക]ഒരു ജീവിയുടെ എല്ലാ കോശങ്ങളും ഒരൊറ്റ കോശത്തിൽ നിന്നാണ് ഉത്ഭവിക്കുന്നത്, അതിനാൽ അവയ്ക്ക് സമാനമായ ജീനോമുകൾ ഉണ്ടായിരിക്കുമെന്ന് പ്രതീക്ഷിക്കുന്നു; എന്നിരുന്നാലും, ചില സന്ദർഭങ്ങളിൽ, വ്യത്യാസങ്ങൾ ഉണ്ടാകുന്നു. കോശവിഭജന സമയത്ത് ഡിഎൻഎ പകർത്തുന്ന പ്രക്രിയയും പരിസ്ഥിതി മ്യൂട്ടജനുകളുമായുള്ള സമ്പർക്കവും സോമാറ്റിക് സെല്ലുകളിൽ മ്യൂട്ടേഷനുകൾക്ക് കാരണമാകും. ചില സന്ദർഭങ്ങളിൽ, അത്തരം മ്യൂട്ടേഷനുകൾ ക്യാൻസറിലേക്ക് നയിക്കുന്നു, കാരണം അവ കോശങ്ങളെ വേഗത്തിൽ വിഭജിക്കുകയും ചുറ്റുമുള്ള ടിഷ്യൂകളെ ആക്രമിക്കുകയും ചെയ്യുന്നു. [35] മനുഷ്യന്റെ രോഗപ്രതിരോധ സംവിധാനത്തിലെ ചില ലിംഫോസൈറ്റുകളിൽ, വി(ഡി)ജെ പുനർസംയോജനം വ്യത്യസ്ത ജനിതക ശ്രേണികൾ സൃഷ്ടിക്കുന്നു, അതായത് ഓരോ കോശവും ഒരു അദ്വിതീയ ആന്റിബോഡി അല്ലെങ്കിൽ ടി സെൽ റിസപ്റ്ററുകൾ ഉത്പാദിപ്പിക്കുന്നു.
ഫിക്ഷനിൽ
[തിരുത്തുക]സയൻസ് ഫിക്ഷൻ കൃതികൾ ജീനോം സീക്വൻസുകളുടെ ലഭ്യതയെക്കുറിച്ചുള്ള ആശങ്കകൾ വ്യക്തമാക്കുന്നു.
മൈക്കൽ ക്രിക്ടന്റെ 1990-ലെ നോവലായ ജുറാസിക് പാർക്കും തുടർന്നുള്ള സിനിമയും, ഒരു വിദൂര ദ്വീപിൽ ക്ലോൺ ചെയ്ത ദിനോസറുകളുടെ തീം പാർക്ക് സൃഷ്ടിക്കുന്ന ഒരു ശതകോടീശ്വരന്റെ കഥ പറയുന്നു. ഒരു ജനിതകശാസ്ത്രജ്ഞൻ പുരാതന കൊതുകുകളുടെ രക്തത്തിൽ നിന്ന് ദിനോസർ ഡിഎൻഎ വേർതിരിച്ചെടുക്കുകയും നിരവധി ഇനം ദിനോസറുകളെ സൃഷ്ടിക്കുകയും ചെയ്യുന്നു. ദിനോസറുകളുള്ള ഒരു ആവാസവ്യവസ്ഥയുടെ എഞ്ചിനീയറിംഗ് സുരക്ഷയെക്കുറിച്ച് തന്റെ വിദഗ്ദ്ധ അഭിപ്രായം നൽകാൻ ഒരു സൈദ്ധാന്തികനോട് ആവശ്യപ്പെടുന്നു, പദ്ധതിയുടെ ഫലങ്ങൾ പ്രവചനാതീതവും ആത്യന്തികമായി നിയന്ത്രണാതീതവുമാകുമെന്ന് അദ്ദേഹം ആവർത്തിച്ച് മുന്നറിയിപ്പ് നൽകുന്നു. ജീനോമിക് വിവരങ്ങൾ ഉപയോഗിക്കുന്നതിന്റെ അപകടങ്ങളെക്കുറിച്ചുള്ള ഈ മുന്നറിയിപ്പുകൾ പുസ്തകത്തിന്റെ ഒരു പ്രധാന വിഷയമാണ്.
1997-ൽ പുറത്തിറങ്ങിയ ഗട്ടാക്ക എന്ന ചലച്ചിത്രം ഒരു ഫ്യൂച്ചറിസ്റ്റ് സമൂഹത്തെ അടിസ്ഥാനമാക്കിയുള്ളതാണ്, അവിടെ അവരുടെ മാതാപിതാക്കളുടെ സ്വഭാവസവിശേഷതകളുടെ ഏറ്റവും അനുയോജ്യമായ സംയോജനം അടിസ്ഥാനമാക്കി കുട്ടികളെ ഉണ്ടാക്കുന്നു. "ഇൻ-വാലിഡ്സ്" എന്നറിയപ്പെടുന്ന യൂജെനിക്സ് പ്രോഗ്രാമിന് പുറത്തുള്ള ആളുകൾ വിവേചനത്തിന് വിധേയരാകുകയും മോശമായ തൊഴിലുകളിലേക്ക് തരംതാഴ്ത്തപ്പെടുകയും ചെയ്യുന്നു. സിനിമയിലെ നായകൻ ഒരു ഇൻ-വാലിഡ് ആണ്, അദ്ദേഹം ഇത്തരം ജനിതക വൈരുദ്ധ്യങ്ങളെ ധിക്കരിക്കുകയും ഒരു ബഹിരാകാശ നാവിഗേറ്ററായി പ്രവർത്തിക്കുക എന്ന തന്റെ സ്വപ്നം സാക്ഷാത്കരിക്കുകയും ചെയ്യുന്നു. ജനിതകമാറ്റം വരുത്തിയ കുട്ടികളും അല്ലാത്തവരും തമ്മിലുള്ള മുൻവിധികളും തീവ്രമായ വർഗവ്യത്യാസങ്ങളും ഉയർത്തുന്ന ഒരു ഭാവിക്കെതിരെ സിനിമ മുന്നറിയിപ്പ് നൽകുന്നു. [36]
ഇതും കാണുക
[തിരുത്തുക]അവലംബം
[തിരുത്തുക]- ↑ Roth, Stephanie Clare (2019-07-01). "What is genomic medicine?". Journal of the Medical Library Association. 107 (3). University Library System, University of Pittsburgh: 442–448. doi:10.5195/jmla.2019.604. ISSN 1558-9439. PMC 6579593. PMID 31258451.
- ↑ 2.0 2.1 2.2 Graur, Dan; Sater, Amy K.; Cooper, Tim F. (2016). Molecular and Genome Evolution. Sinauer Associates, Inc. ISBN 9781605354699. OCLC 951474209.
- ↑ Brosius, J (2009). "The Fragmented Gene". Annals of the New York Academy of Sciences. 1178 (1): 186–93. Bibcode:2009NYASA1178..186B. doi:10.1111/j.1749-6632.2009.05004.x. PMID 19845638.
- ↑ "The Human Genome Project". Genome.gov. Retrieved 2023-04-29.
- ↑ "First complete sequence of a human genome". National Institutes of Health (NIH). 2022-04-11. Archived from the original on 2023-04-14. Retrieved 2023-04-29.
- ↑ Hartley, Gabrielle (31 March 2022). "The Human Genome Project pieced together only 92% of the DNA – now scientists have finally filled in the remaining 8%". TheConversation.org. The Conversation US, Inc. Retrieved 4 April 2022.
- ↑ Winkler HL (1920). Verbreitung und Ursache der Parthenogenesis im Pflanzen- und Tierreiche. Jena: Verlag Fischer.
- ↑ "definition of Genome in Oxford dictionary". Archived from the original on 1 March 2014. Retrieved 25 March 2014.
- ↑ "genome". Oxford English Dictionary (Online ed.). Oxford University Press. (Subscription or participating institution membership required.)
- ↑ Lederberg, Joshua; McCray, Alexa T. (2001). "'Ome Sweet 'Omics – A Genealogical Treasury of Words" (PDF). The Scientist. 15 (7). Archived from the original (PDF) on 29 September 2006.
- ↑ "The ingenuity of bacterial genomes". Annual Review of Microbiology. 74: 815–834. 2020. doi:10.1146/annurev-micro-020518-115822. PMID 32692614.
- ↑ Brown, TA (2018). Genomes 4. New York, NY, USA: Garland Science. ISBN 9780815345084.
- ↑ "Ensembl Human Assembly and gene annotation (GRCh38)". Ensembl. Retrieved May 30, 2022.
- ↑ "All about genes". beowulf.org.uk.
- ↑ "Genome Home". 2010-12-08. Retrieved 27 January 2011.
- ↑ Zimmer C (18 December 2013). "Toe Fossil Provides Complete Neanderthal Genome". The New York Times. Archived from the original on 2022-01-02. Retrieved 18 December 2013.
- ↑ Prüfer K, Racimo F, Patterson N, Jay F, Sankararaman S, Sawyer S, et al. (January 2014). "The complete genome sequence of a Neanderthal from the Altai Mountains". Nature. 505 (7481): 43–49. Bibcode:2014Natur.505...43P. doi:10.1038/nature12886. PMC 4031459. PMID 24352235.
- ↑ Wade N (2007-05-31). "Genome of DNA Pioneer Is Deciphered". The New York Times. Retrieved 2 April 2010.
- ↑ "What's a Genome?". Genomenewsnetwork.org. 2003-01-15. Retrieved 27 January 2011.
- ↑ "Mapping Factsheet". 2004-03-29. Archived from the original on 19 July 2010. Retrieved 27 January 2011.
- ↑ Gelderblom, Hans R. (1996). Structure and Classification of Viruses (4th ed.). Galveston, TX: The University of Texas Medical Branch at Galveston. ISBN 9780963117212. PMID 21413309.
- ↑ "Archaeal chromosome biology". Journal of Molecular Microbiology and Biotechnology. 24 (5–6): 420–27. 2014. doi:10.1159/000368854. PMC 5175462. PMID 25732343.
- ↑ Chaconas, George; Chen, Carton W. (2005). "Replication of Linear Bacterial Chromosomes: No Longer Going Around in Circles". The Bacterial Chromosome. pp. 525–540. doi:10.1128/9781555817640.ch29. ISBN 9781555812324.
- ↑ "Bacterial Chromosomes". Microbial Genetics. 2002.
- ↑ "Constraints and plasticity in genome and molecular-phenome evolution". Nature Reviews. Genetics. 11 (7): 487–98. July 2010. doi:10.1038/nrg2810. PMC 3273317. PMID 20548290.
- ↑ "Extreme genome reduction in symbiotic bacteria". Nature Reviews. Microbiology. 10 (1): 13–26. November 2011. doi:10.1038/nrmicro2670. PMID 22064560.
- ↑ "Insights from 20 years of bacterial genome sequencing". Functional & Integrative Genomics. 15 (2): 141–61. March 2015. doi:10.1007/s10142-015-0433-4. PMC 4361730. PMID 25722247.
- ↑ "Scientists sequence asexual tiny worm whose lineage stretches back 18 million years". ScienceDaily. Retrieved 7 November 2017.
- ↑ Khandelwal, Sharda (March 1990). "Chromosome evolution in the genus Ophioglossum L.". Botanical Journal of the Linnean Society. 102 (3): 205–17. doi:10.1111/j.1095-8339.1990.tb01876.x.
- ↑ 30.0 30.1 Zhou, Wanding; Liang, Gangning; Molloy, Peter L.; Jones, Peter A. (11 August 2020). "DNA methylation enables transposable element-driven genome expansion". Proceedings of the National Academy of Sciences of the United States of America. 117 (32): 19359–19366. Bibcode:2020PNAS..11719359Z. doi:10.1073/pnas.1921719117. ISSN 1091-6490. PMC 7431005. PMID 32719115.
- ↑ 31.0 31.1 Lewin, Benjamin (2004). Genes VIII (8th ed.). Upper Saddle River, NJ: Pearson/Prentice Hall. ISBN 978-0-13-143981-8.
- ↑ Stojanovic, Nikola, ed. (2007). Computational genomics : current methods. Wymondham: Horizon Bioscience. ISBN 978-1-904933-30-4.
- ↑ Nurk, Sergey; et al. (2022-03-31). "The complete sequence of a human genome" (PDF). Science. 376 (6588): 44–53. Bibcode:2022Sci...376...44N. doi:10.1126/science.abj6987. PMC 9186530. PMID 35357919. Archived from the original (PDF) on 2022-05-26.
- ↑ "Eukaryotic genome size databases". Nucleic Acids Research. 35 (Database issue): D332–38. January 2007. doi:10.1093/nar/gkl828. PMC 1669731. PMID 17090588.
- ↑ "Somatic mutation in cancer and normal cells". Science. 349 (6255): 1483–89. September 2015. Bibcode:2015Sci...349.1483M. doi:10.1126/science.aab4082. PMID 26404825.
- ↑ "Gattaca (movie)". Rotten Tomatoes. 24 October 1997.
കൂടുതൽ വായനയ്ക്ക്
[തിരുത്തുക]- Benfey P, Protopapas AD (2004). Essentials of Genomics. Prentice Hall.
- Brown TA (2002). Genomes 2. Oxford: Bios Scientific Publishers. ISBN 978-1-85996-029-5.
- Gibson G, Muse SV (2004). A Primer of Genome Science (Second ed.). Sunderland, Mass: Sinauer Assoc. ISBN 978-0-87893-234-4.
- Gregory TR (2005). The Evolution of the Genome. Elsevier. ISBN 978-0-12-301463-4.
- Reece RJ (2004). Analysis of Genes and Genomes. Chichester: John Wiley & Sons. ISBN 978-0-470-84379-6.
- Saccone C, Pesole G (2003). Handbook of Comparative Genomics. Chichester: John Wiley & Sons. ISBN 978-0-471-39128-9.
- Werner E (December 2003). "In silico multicellular systems biology and minimal genomes". Drug Discovery Today. 8 (24): 1121–27. doi:10.1016/S1359-6446(03)02918-0. PMID 14678738.
പുറം കണ്ണികൾ
[തിരുത്തുക]- UCSC ജീനോം ബ്രൗസർ - 80-ലധികം ജീവികളുടെ ജീനോമും വ്യാഖ്യാനങ്ങളും കാണുക.
- genomecenter.howard.edu (ആർക്കൈവ് ചെയ്തത് 9 ഓഗസ്റ്റ് 2013)
- ഒരു ഡിഎൻഎ തന്മാത്ര നിർമ്മിക്കുക (ആർക്കൈവ് ചെയ്തത് 9 ജൂൺ 2010)
- ചില താരതമ്യ ജീനോം വലുപ്പങ്ങൾ
- ഡിഎൻഎ ഇന്ററാക്ടീവ്: ഡിഎൻഎ ശാസ്ത്രത്തിന്റെ ചരിത്രം
- തുടക്കം മുതൽ ഡിഎൻഎ
- ഹ്യൂമൻ ജീനോം പ്രോജക്റ്റിനെക്കുറിച്ച് എല്ലാം - Genome.gov-ൽ നിന്ന്
- അനിമൽ ജീനോം സൈസ് ഡാറ്റാബേസ്
- പ്ലാന്റ് ജീനോം സൈസ് ഡാറ്റാബേസ് (ആർക്കൈവ് ചെയ്തത് 1 സെപ്റ്റംബർ 2005)
- GOLD:Genomes ഓൺലൈൻ ഡാറ്റാബേസ്
- ജീനോം ന്യൂസ് നെറ്റ്വർക്ക്
- NCBI എൻട്രസ് ജീനോം പ്രോജക്റ്റ് ഡാറ്റാബേസ്
- NCBI ജീനോം പ്രൈമർ
- ജീൻകാർഡുകൾ - മനുഷ്യ ജീനുകളുടെ ഒരു സംയോജിത ഡാറ്റാബേസ്
- ബിബിസി ന്യൂസ് – അന്തിമ ജീനോം 'അധ്യായം' പ്രസിദ്ധീകരിച്ചു
- IMG (ഇന്റഗ്രേറ്റഡ് മൈക്രോബയൽ ജീനോംസ് സിസ്റ്റം) - DOE-JGI യുടെ ജീനോം വിശകലനത്തിനായി
- GeKnome ടെക്നോളജീസ് നെക്സ്റ്റ്-ജെൻ സീക്വൻസിംഗ് ഡാറ്റാ അനാലിസിസ് —ഇല്ലുമിനയ്ക്കായുള്ള അടുത്ത തലമുറ സീക്വൻസിംഗ് ഡാറ്റ വിശകലനം, GeKnome ടെക്നോളജീസിൽ നിന്നുള്ള 454 സേവനങ്ങൾ (ആർക്കൈവ് ചെയ്തത് 3 മാർച്ച് 2012)
